Сплайсинг
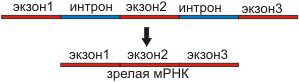 Схема сплайсинга
Схема сплайсинга
Многие гены состоят из экзонов - кодирующие участки и интронов – некодирующие участки. При транскрипции с гена считывается РНК несущая как экзоны, так и интроны. В процессе сплайсинга интроны вырезаются, а экзоны сшиваясь образуют зрелую РНК.
Характеристика экзонов и интронов некоторых генов.
| продукт гена | организм | длина экзонов, пн |
интроны
|
|
| число | общая длина, пн | |||
| аденозиндезаминаза | человек | 1500 | 11 | 30000 |
| аполипротеин B | человек | 14000 | 28 | 29000 |
| β-глобин | мышь | 432 | 2 | 762 |
| цитохром b | митохондрии дрожжей | 2200 | 6 | 5100 |
| дигидрофолатредуктаза | мышь | 568 | 5 | 31500 |
| эритропоэтин | человек | 582 | 4 | 1562 |
| фактор V[1] | человек | 9000 | 25 | 177000 |
| фиброин шелка | шелкопряд | 18000 | 1 | 970 |
| гипоксантин-фосфорибозилтрансфераза | мышь | 1307 | 8 | 32000 |
| α-интерферон | человек | 600 | 0 | 0 |
Сплайсосомы
У эукариот в большинстве кодирующих белки РНК интроны вырезаются в сплайсосомах - комплексах, состоящих из пяти малых ядерных нуклеопротеинов (snRNP, или SNURPs - снурпс). Интроны содержат последовательности, необходимые для вырезания - 3'-сайт (акцепторный), 5'-сайт (донорный) и бранч-сайт (см. рис.2). РНК в snRNA взаимодействует с этими сайтами, способствуя вырезанию интронов.

рис.2 Консервативные последовательности в экзонах и интронах необходимые для сплайсинга.
Обнаружено два типа сплайсосом - большие и малые, состоящие из разных snRNP.
Большие сплайсосомы состоят из U1, U2, U4, U5 и U6 snRNP; транскрипция U1-U5 РНК осуществляется РНК-полимеразой II, а U6 - РНК полимеразой III. Вырезают интроны имеющие GU на 5'-сайте и AG на 3'-сайте. U1 связываются с 5'-сайтом, U2 связываются с бранч-сайтом и U6. U4 ингибирует U6, инактивируя сплайсосому. U5 связывает U1 и U2 при образовании 'лассо'. При активации U6, U1 перемещается и связывает U2. U2-U6 формируют активный католитический комплекс (см. рис.3).
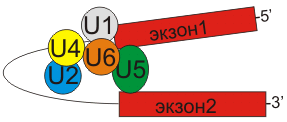
рис.3 Сплайсосома и взаимодействие ее компонентов с экзонами и интронами РНК.
Малые сплайсосомы. Сплайсируют РНК, содержащие AU 3'- и AC 5'-концы интронов. Cостоят из U11, U12, U4atac, U6atac и U5 - одинакового для больших и малых сплайсосом.
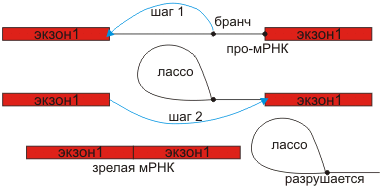
рис.4 Механизм удаления интронов.
Малые РНК. sРНК 103-105 копий на клетку, обогащены урацилом U1, U2... 100-300н, кодируются в ядре, но работают как в ядре (small nuclear - SN), так и в цитоплазме (small cytoplasmic - SC)
SNURPS - РНП (рибонуклеопротеидные комплексы) в ядре. snРНК входят в состав РНП, участвующих в полиаденилированиии и сплайсинге | SCURPS - РНП в цитоплазме, входят в состав информосом | U4 в комплексах, участвующих в полиаденилировании (при красной волчанке (аутоимунном заболевании) вырабатываются антитела к белкам комплекса с U4 нет полиаденилирования и сплайсинга) | Гистоновая mРНК не полиаденилируется т.к sРНКU7, комплементарная 3'-концу гистоновой mРНК, защищает ее от полиаденилирования.
Сплайсинг может иметь позитивную и негативную регуляцию. При позитивной регуляции из первичного транскрипта вырезается интрон при действии белка активатора. При негативной первичный транскрипт не подвергается сплайсингу при действии
белка репрессора.
Транс-сплайсинг
Транс-сплайсинг - форма
сплайсинга, при которой соединяются РНК разных транскриптов.
Альтернативный сплайсинг
mРНК кальцитонинового гена у млекопитающих (крыса)
Во всех клетках есть кальцитониновый ген, но в клетках щитовидной железы он экспрессируется в виде гормона кальцитонина, а в клетках гипофиза - нейропептида CGRP (пептида, имеющего отношение к гену кальцитонина). Ген один, а белки получаются разные в результате сплайсинга mРНК и процессинга полипептидов. В клетках других тканей этот ген не экспрессируется.
Сплайсинг осуществляется белковыми комплексами – сплайсосомами-ферменты, вырезающие и сшивающие участки про-mРНК, белки, придающие про-mРНК нужную конформацию, sPНК. Сплайсосома связана с ферментами полиаденилирования.
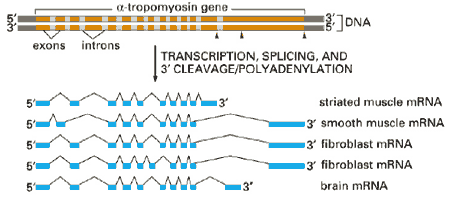
рис. Схема образования различных мРНК из
одного тропомиозинового гена в различных клетках.
Автосплайсинг
Автосплайсинг - вид сплайсинга, когда интроны кодируют рибозимы - РНК с каталитической активностью, выполняющие роль сплайсосомы. Имеются три группы интронов кодирующих рибозимы Group I, II и III. Group II и III выполняют роль сплайсосомы без привлечения
других белков.
Автосплайсинг открыт Томасом Чеком (США) в 1982 году. Он работал с инфузорией
Tetrаchymenа thermophyla
. У этой инфузории образуется 35S про-rРНК длиной 6400 нуклеотидов. Без участия дополнительных соединений белковой природы из этой про-rРНК вырезается внутренний участок длиной в 414 нуклеотида. Два экзона сшиваются с образованием 26S rРНК. Единственное требование - определенная концентрация ионов магния. Про-rРНК имеет третичную структуру и обладает каталитичекой активностью. Впервые было показано, что каталитической активностью
обладают не только белки.
Small nuclear RNAs (snRNAs), ranging in size from about 80 to 350 nucleotides, are ubiquitous components of eukaryotic cells. The U (uridine-rich) family of snRNAs are thought to be important for RNA splicing and have been highly conserved in evolution. They are associated with specific polypeptides, forming small nuclear ribonucleoprotein complexes (snRNPs). These are often targets of the autoimmune disorder systemic lupus erythematosus. The polypeptide associated with U1 snRNA appears to be unique (Wooley et al., 1983). Multigene families for human U1, U2, U3, U4 and U6 snRNAs have been demonstrated. Transcription of U1-U5 RNAs is accomplished by RNA polymerase II, whereas U6 snRNA is thought to be transcribed by RNA polymerase III. Kunkel and Pederson (1988) studied the upstream regulatory elements for the human U6 RNA gene and found a marked similarity to the proximal control elements of U1 and U2 snRNA genes, despite the transcription by different polymerases. Most of the human genes complementary to these snRNAs are pseudogenes, which are dispersed in the genome. Although most of the U1 genes are on chromosome 1, they probably are separated by intergenic spacer regions larger than 15 kb, because none of the recombinant phages isolated to date contains more than one U1 gene. By way of contrast, U2 snRNA genes are organized as a nearly perfect tandem array of 10 to 20 copies per haploid genome (Van Arsdell and Weiner, 1984). Bostock et al. (1984) concluded that the genes for human U1 snRNA are clustered on the short arm of chromosome 1. By somatic cell hybrid studies, Naylor et al. (1984) found that RNU1 segregated with PEPC (170000) and AK2 (103020), chromosome 1 markers. By in situ hybridization, they showed that most of the grains were concentrated in band 1p36.3. By in situ hybridization, Lindgren et al. (1985) found that the U1 snRNA pseudogenes (called class I) are coded in a cluster in 1q12-q22, separate from the true genes (in about 30 copies) in 1p36. Bernstein et al. (1985) presented evidence in support of the idea that the true U1 genes were derived by gene amplification and transposition from a more ancient family of U1 genes (represented now by class I U1 pseudogenes). The clustering of both U1 true genes and pseudogenes and the conservation of at least 44 kb of DNA flanking the U1 coding region in a large fraction of the 30 true U1 genes
are explained by gene amplification.
Pseudogenes for U1 snRNA outnumber the true genes by 15- to 30-fold. Some of the pseudogenes have no flanking homology to the true genes, but others, the class I pseudogenes, share several kilobases of flanking homology. Lindgren et al. (1985) noted that the site of the U1 pseudogenes corresponds to a site of chromosomal modification by adenovirus-12. They postulated that class I U2 pseudogenes may be affected by the virus because they retain flanking regulatory sequences.
